Detta är en kort introduktion till linjär regressionsanalys i R. Texten förutsätter att du förstår metodernas matematik och logik. Se även min korta introduktion till R, min superkorta introduktion till datatabeller i R och min ultrakorta introduktion till diagram i R.
1. Vi kan estimera en linjär regressionsmodell med minstakvadratmetoden i R med funktion lm(). Här är ett exempel där vi använder de två argumenten formula och data i parentesen till lm():
# skapa en tabell min_tabell <- tibble(
y=c(2,5,7,8), x=c(3,5,4,6))
| y | x |
| 2 | 3 |
| 5 | 4 |
| 7 | 5 |
| 8 | 6 |
# Estimera regressionsmodellenlm(formula = y~x , data= min_tabell)
Om vi kör koden ovan visas i konsolen estimaten för de två koefficienterna a och b från regressionsmodellen där y och x är variabler och u är feltermen:

2. Funktionen lm() skapar flera resultat. Ett sätt att se dessa är att skicka resultatet av lm() till funktionen summary(). Exempel:
lm(formula = y~x , data= min_tabell) %>% summary()
I konsolen (console) får vi nu den typ av resultat som är vanliga för denna typ av funktioner i statistiska analysprogram. Bland annat ett t-test per konstant i regressionsmodellen samt ett resultat av ett F-test för hela regressionsmodellen.

3. Ett annat sätt att nå de saker som funktion lm() skapar är att spara resultatet i ett nytt objekt.
reg_resultat <- lm(formula = y~x , data= min_tabell)
Ett sätt att få en överblick över innehållet är att använda funktionen head():
reg_resultat %>% head()
Funktionen head() visar de första delarna av ett objekt. Om objektet är en tabell visas de översta raderna. I detta fall innehåller vårt objekt olika delar. Det objekt som funktion lm() skapar innehåller olika delar. Funktionen head() innebär att vi får se de första observationerna i varje sådan del. Bland annat:
$residuals
1 2 3 4
-1.1 -1.3 2.3 0.1
$effects
(Intercept) x
-11.000000 -3.577709 2.666667 1.043498
4. Enskilda variabler, delar, av lm-objektet kan hämtas med hjälp av dollartecken. Exempel:
# Returnera predikterade värden,
reg_resultat$fitted.values
# Returnera residualer,
reg_resultat$residuals
5. Delar av lm-objektet kan hämtas med särskilda funktioner. Exempel:
# Estimerade koefficienterna
reg_resultat %>% coef()
# Predikterade värden
reg_resultat %>% predict()
# Residualerna
reg_resultat %>% residuals()
6. Vi kan rita ut datapunkterna för två variabler och regressionslinjen med hjälp av ggplot() och geom_smooth(). Vi börjar med att skapa en textvariabel med värdena för x respektive y per punkt, med hjälp av funktionen paste(). Vi använder geom_label() för att rita ut punkternas x- och y-värden, genom att hämta dem från den nya variabel vi skapade med paste(). Exempel:
min_tabell %>%
mutate(labelvar=paste(x,y)) %>%
ggplot(aes(x=x, y=y, label=labelvar)) +
geom_label(vjust=-.5) +
ylim(2,9) +
geom_point(size=4) +
geom_smooth(method="lm", se=FALSE)
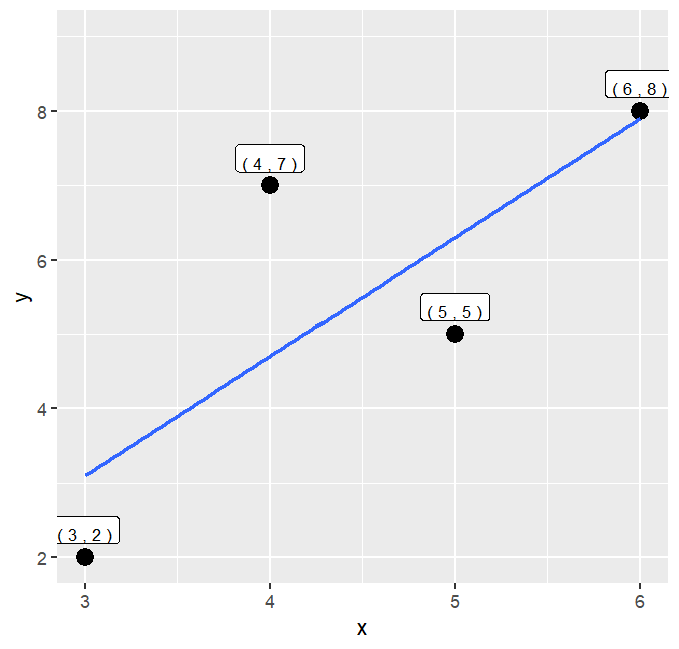
Alternativet se=FALSE innebär att diagrammet inte visar konfidensintervall. Definiera detta alternativ som se=TRUE för att be programmet rita ut konfidensintervallet i diagrammet.
7. För att granska residualerna kan vi hämta värdena från det objekt som funktion lm() skapar. En annan metod är att använda funktionen autoplot() från paketet ggfortify. Funktion autoplot() ritar bland annat ut 6 olika diagram Exempel:
install.packages("ggfortify") # körs en gång
library("ggfortify") # körs varje gång vi startar om R
reg_resultat %>% autoplot()

Lägger vi till alternativet which=c(5,6) får vi de två sista diagrammen, nr 5 respektive 6.
reg_resultat %>% autoplot(which=c(5,6))

8. Vi kan skapa dummyvariabler när vi estimerar regressionsmodellen, med hjälp av funktionen factor(). Vi har följande tabell:
| y | x | k |
| 2 | 3 | 0 |
| 5 | 5 | 0 |
| 7 | 4 | 1 |
| 8 | 6 | 1 |
# spara tabellen som objektmin_tabell_2 <- tibble(
y=c(2,5,7,8),
x=c(3,5,4,6),
k=c(0,0,1,1))
Nu ska vi estimera regressionsmodellen där
och
är konstanter,
och
är variabler, samt
är felterm. För att vara säker på att programmet förstår att
är en dummyvariabel använder vi funktionen
factor():
lm(y ~ x + factor(k) , data=min_tabell_2)
9. Använd interaktionstermer genom att multiplicera variablerna i parentesen till lm(). Här följer ett exempel där vi använder samma tabell som i föregående punkt.
Vi ska nu estimera regressionsmodellen där
är konstanter, variablerna är desamma som i föregående exempel och
är felterm.
lm(y ~ x*factor(k), data=min_tabell)
Resultat:
Coefficients:
(Intercept) x factor(k)1 x:factor(k)1
-2.5 1.5 7.5 -1.0
I just dessa exempel använder vi en variabel som redan är kodad som en dummyvariabel med värdena 0 och 1. I just detta fall får vi samma resultat om vi utelämnar funktion factor() vid estimeringen av regressionsmodellerna.
10. För att använda minstakvadratmetoden med robusta standardfel kan vi använda funktionen lm_robust() från paket estimar. Här är ett exempel där vi estimerar regressionsmodellen (samma som ovan) med Whites robusta standardfel version HC0 och HC1:
lm_robust(y ~x , data=min_tabell, se_type="HC0")
lm_robust(y ~x , data=min_tabell, se_type="HC1")
Läs mer om lm_robust här. Läs mer om paketet estimar här. Det finns även andra kommandon som ger samma resultat.
11. För att skapa snygga resultattabeller kan vi använda funktionen stargazer() från paketet stargazer. Stargazer kan bland annat skapa resultattabeller i tex-format och exportera dessa till filer på hårddisken. Stargazer funkar direkt på resultat från lm(). Exempel:
install.packages("stargazer") # körs en gång
library("stargazer")
lm(y ~ x , data=min_tabell) %>% stargazer(type="text")

Här finns en guide för hur vi med hjälp av funktionen starprep() kan kombinera lm_robust() med stargazer().
12. Det finns många andra färdiga funktioner i R som är användbara för regressionsanalys. Se till exempel denna lista.